2026年1月3日,生物信息领域重要期刊International Journal of Biological Macromolecules在线发表了ios版雷竞技官网智慧农业学院(人工智能学院)计智伟教授课题组的题为“MsipNet: a multi-scale representation learning framework for predicting protein-RNA interaction”的研究论文。在这项工作中,研究人员开发了一个多尺度表示学习框架MsipNet,用于预测RNA结合蛋白(RBP)的结合偏好及其靶向RNA。

据悉,该团队成功研发了一个名为MsipNet的多尺度表示学习框架,可实现对蛋白质-RNA相互作用的高精度预测(图1)。这个计算框架首先通过多模态表示学习模块,整合RNA序列的全局和局部特征并融合RNA结构信息(基于icSHAPE数据),形成全面的特征表示;随后,借助LSTM与UCDC(U形卷积-空洞卷积)网络的协同优化,对多尺度特征进行深层次提炼与融合;最终输出准确的RNA结合偏好预测结果。该设计以模块化、连贯的架构突出了多尺度特征学习在提升预测性能中的关键作用,为RNA功能机制解析及相关药物设计提供了高效计算工具。
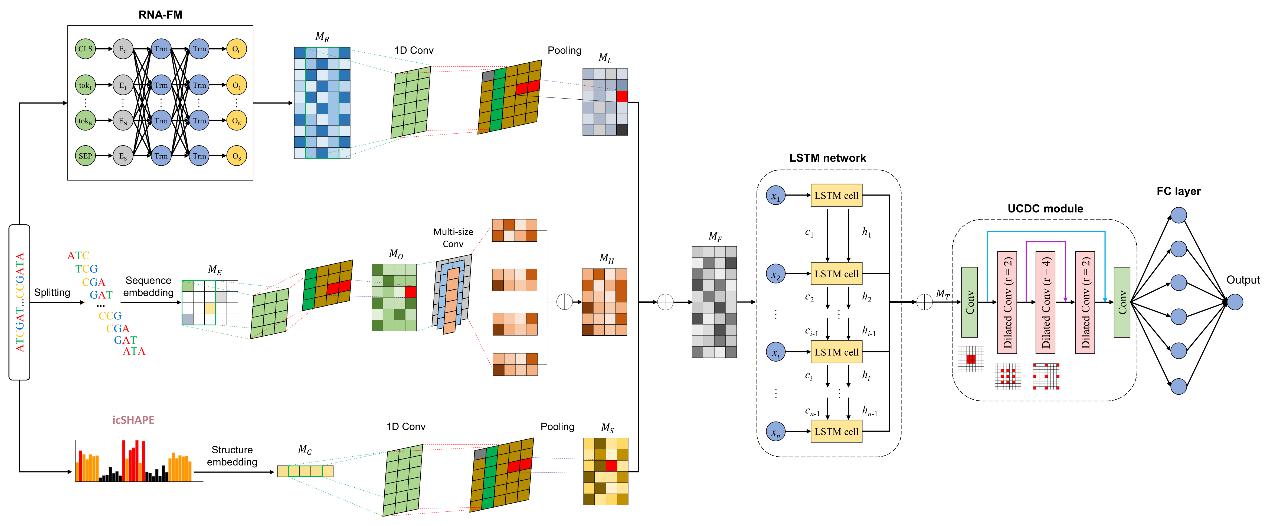
图1. MsipNet算法框架图
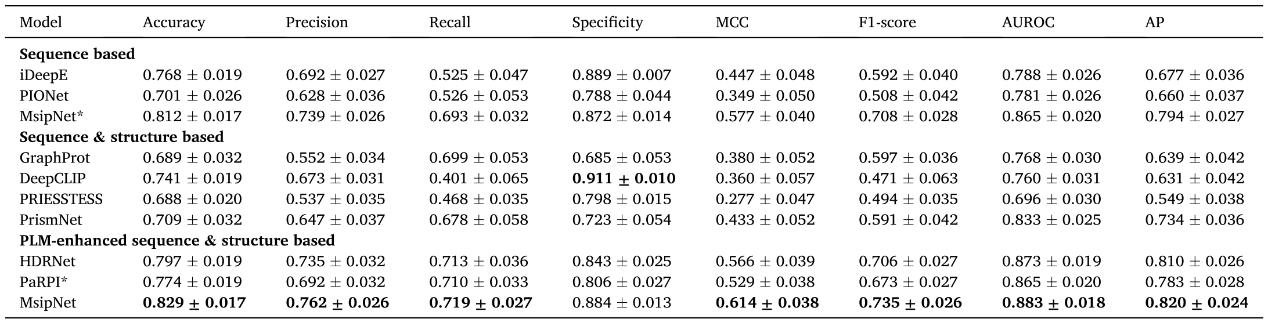
在性能评估中,MsipNet与八种现有计算模型进行了系统对比。结果表明,MsipNet取得了最优的预测性能,其AUROC达到0.883,显著优于所有现有方法。即便忽略结构数据(icSHAPE),其表现也明显超过同类基于序列的方法。在其余对比模型中,表现最接近的HDRNet(AUROC=0.873)和PrismNet(AUROC=0.833)仍与MsipNet存在明显差距;而传统方法如DeepCLIP等的预测精度下降更为显著。此外,在不同正负样本比例的不平衡数据条件下,MsipNet同样展现出更稳健的性能优势。这些结果一致表明,MsipNet所采用的多尺度表示学习框架具有显著优越性,为蛋白质-RNA相互作用预测设立了新的精度标杆。
为深入揭示MsipNet优越性能的内在机理,研究团队对模型识别出的RNA序列motif进行了可视化分析。结果表明,MsipNet在motif发现能力上显著优于其他模型,所识别出的13个motif与权威数据库CisBP-RNA中的已知motif高度吻合,尤其擅长捕捉G-rich、U-rich等具有特定结构的基序模式(图2)。
更重要的是,为验证所发现motif的生物学真实性,作者进一步将上述13个motif与另一实验验证数据库ATtRACT进行了交叉比对。结果显示,MsipNet所预测的motif获得了压倒性的实验数据支持:在CisBP-RNA匹配的motif中,有8个同样存在于ATtRACT的实验(如RNAcompete或SELEX)数据中,这一验证率远高于其他对比模型。这充分证明,MsipNet不仅是一个高精度的预测工具,更能可靠地发现具有真实生物学功能的RNA结合motif,为探索蛋白质-RNA相互作用的分子机制提供了直接线索。
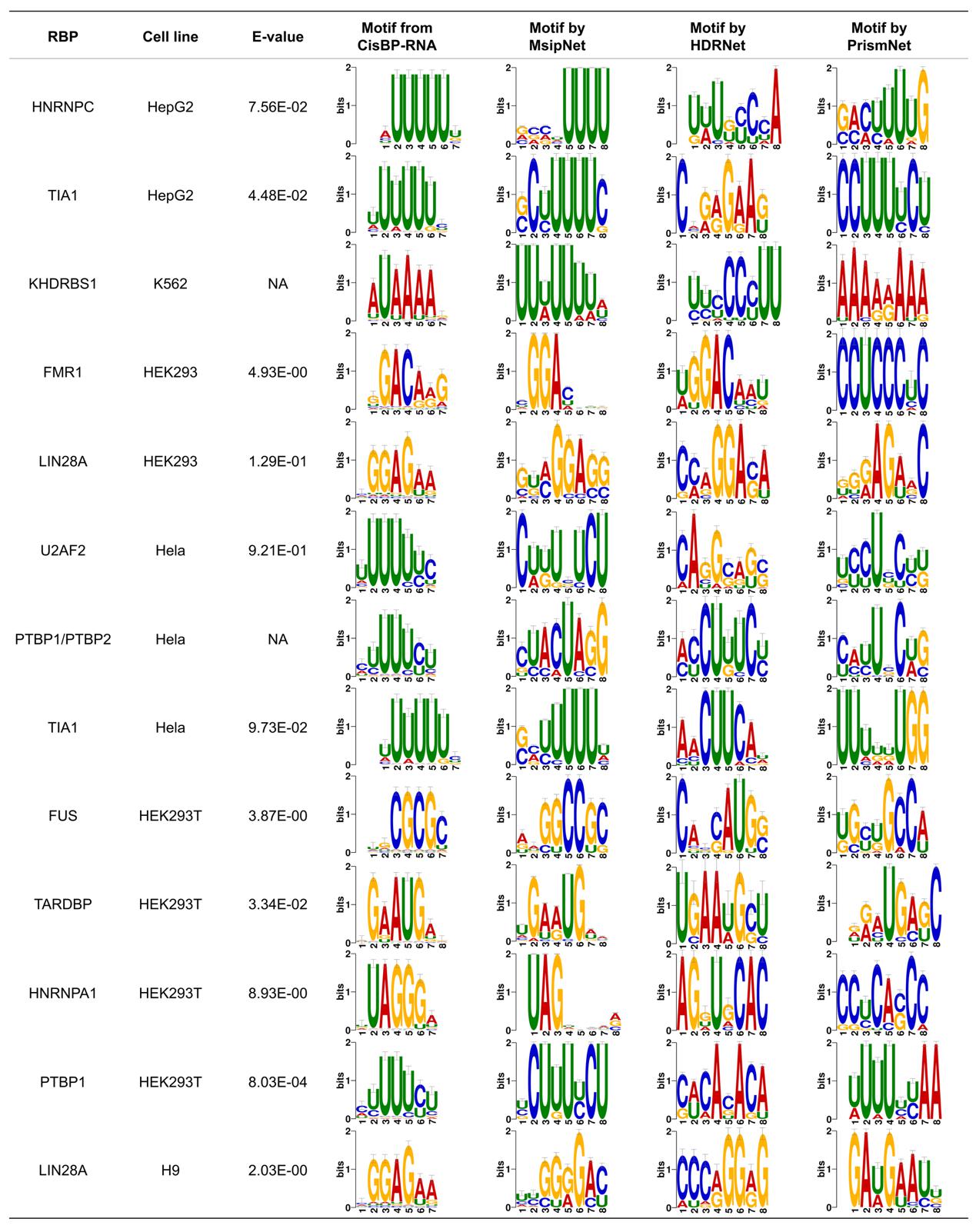
图2. MsipNet在42个RBP数据集中识别出13个经数据库报道的RNA结合motif
综上所述,本研究提出的多尺度表示学习框架MsipNet,通过整合RNA序列的多模态特征与结构信息,并结合LSTM与UCDC网络进行层级特征提炼,实现了对蛋白质-RNA相互作用的高精度、高泛化能力预测。该模型在多项基准测试中显著优于现有方法,不仅能稳定、高效地识别结合位点,还展现出卓越的生物学可解释性,成功发掘出多组经实验验证的RNA结合基序。这些成果表明,MsipNet不仅为蛋白质-RNA相互作用预测设立了新的技术标准,也为深入解析RNA调控机制及发现潜在药物靶点提供了可靠的计算工具。
本文的第一作者为ios版雷竞技官网智慧农业学院(人工智能学院)2023级硕士生宋楠,通讯作者为计智伟教授。中国科学技术大学李志锦博士、哈尔滨工业大学邓阳博士和ios版雷竞技官网黄金虎副教授参与了这项工作。UNC Chapel Hill的Weiling Zhao教授为论文的撰写提供了宝贵建议。本项工作受到2025年ios版雷竞技官网“滨江基石”交叉专项、江苏省农业自主创新项目等经费支持。
原文链接:https://doi.org/10.1016/j.ijbiomac.2025.149883
阅读次数:17
【 转载本网文章请注明出处 】





